Anforderungsformulare
Hier finden sie alle nötigen Formulare für die Anforderung einer ESR1 liquid biopsy MutationsanalyseAbnahme von humanem Vollblut in Cell FreeDNA Collection Tubes zur ESR1-Testung an Liquid-Biopsies
Checkliste für den Versand
Kostenübernahmeerklärung für privat versicherte Patienten
ESR1 im Mammakarzinom
ER-positive, HER2-negative Mammakarzinome sprechen in der Regel auf eine antihormonelle Therapie mit Aromataseinhibitoren (AI) an. Bis zu 40 Prozent der hiermit behandelten Patienten entwickeln jedoch eine durch ESR1-Mutationen ausgelöste Resistenz gegen diese endokrine Standardtherapien. Aktivierende Mutation in der ESR1-Ligandenbindedomäne bewirken eine ligandenunabhängige konstitutive ESR1-Aktivierung welche zu einer Resistenz gegen führen.
Patienten, bei denen eine solche ESR1-Variante
vorliegt, können nun jedoch von einer zielgerichteten Behandlung mit
Elacestrant (Handelsname Orserdu) profitieren. Elacestrant ist ein
selektiver östrogen-Rezeptor-Degrader (SERD), der gezielt an
östrogenrezeptor-Proteine mit einer aktivierenden ESR1-Variante
bindet und diese degradiert, so dass keine Wachstumssignale mehr
über den Rezeptor gesendet werden können.
Elacestrant kann bei Patienten eingesetzt werden, deren Mammakarzinom
eine Resistenz gegen endokrine Standardtherapien, auch unter Verwendung eines
CDK-4/6-Inhibitors, entwickeln.
ESR1 Struktur und Mutationen
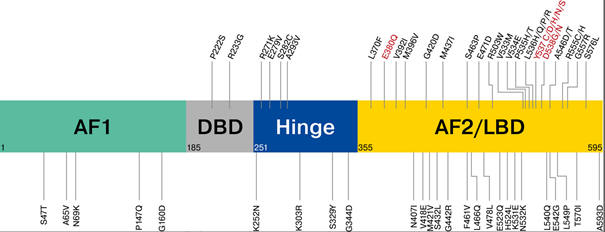
Quelle: https://doi.org/10.1002/cncr.32345
Aktivierende,
klinisch signifikante Mutationen zeigen sich in der AF2/LBD
(Activation function-2 / Ligand Binding Domain), welche von den
Kodons 355 bis 596 reicht.
Die aktivierenden Mutationen
konzentrieren sich stark auf zwei Hotspots um die Kodons 380 und
537/538:
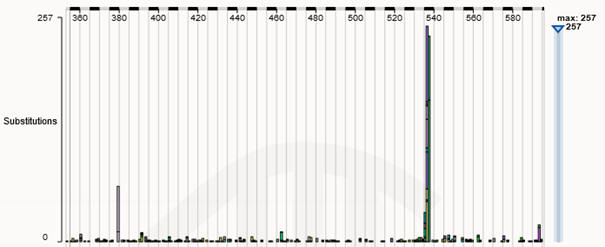
Unser dPCR-Assay erfasst folgende 8 aktivierende
ESR1-Mutationen der AF2/LBD-Domäne:
E380Q, D538G, L536H, Y537C,
L536P, Y537N, L536R, Y537S
Anzahl der in COSMIC dokumentierten Alterationen mit Effekt auf eine Aminosäure in der
AF2/LBD-Domäne mit >4 Einträgen: 661
Anzahl der von unserem
Assay erfassten COSMIC Mutationen: 593 (89,7 % der Mutationen)
Erfassung der COSMIC-Mutationen und deren Häufigkeit im Detail

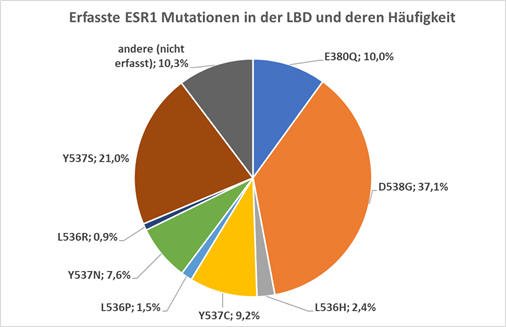
Unser dPCR-Assay deckt 90% der signifikanten,
aktivierenden ESR1 Mutationen der AF2/LBD-Domäne ab.
Methodik
cfDNA (circulating free DNA)-Isolation aus Liquid
Biopsies
cfDNA kommt natürlicher Weise in geringen
Konzentrationen u. a. im Blut vor und stammt von z. B. apoptotisch
zugrunde gegangenen Zellen. Der größte Teil der cfDNA ist in 165bp
oder einem Mehrfachen von 165bp fragmentiert, was die Nukleosomen
(DNA-Histon-Komplex) widerspiegelt.
Die cfDNA aus nekrotischen Zellen kann aus weit größeren Fragmenten bestehen.
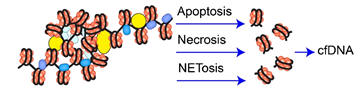
cfDNA kann DNA verschiedener Ursprünge enthalten: circulating tumor DNA
(ctDNA), cell-free mitochondrial DNA (ccf mtDNA), cell-free fetal
DNA (cffDNA) u. a.
Das Target der Molekularpathologie ist natürlich die circulating tumor DNA = ctDNA, deren Konzentration im
Blut von vielen Faktoren wie Tumormasse, Zustand und Therapie des
Patienten, etc. beeinflusst wird.
Eine valide Untersuchung der ctDNA wird eine gewisse analytische Herausforderung im Bereich
der Spurenanalytik.
Obwohl zur Probenahme spezielle Blutröhrchen mit einer Stabilisierungslösung verwendet werden sollten die Proben
möglichst frisch sein, keinen warmen Temperaturen und keinem
mechanischen Stress, z. B. starken Erschütterungen ausgesetzt sein.
Zur Probenahme sollten keine dünnen Kanülen verwendet werden, um
ein Scheren der DNA-haltigen Leukozyten zu vermeiden, was die ctDNA
stark mit Leukozyten-DNA verdünnen würde.
Im Labor werden die Blutproben relativ lange und sanft zentrifugiert. Das separierte
Plasma wird abgenommen und in spezielle, aufwendige
Isolationsverfahren für ctDNA eingesetzt.
Auch im Optimalfall sind die Ausbeuten der ctDNA im Vergleich zu DNA aus Geweben
üblicherweise sehr gering und im Nanogrammbereich.
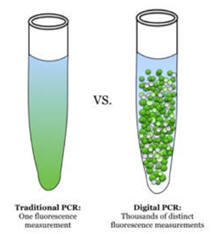
Digitale PCR (pPCR)
Bei der dPCR wird eine möglichst große Menge
an DNA-Isolation in allelspezifische PCRs eingesetzt, deren
jeweilige Positivität durch eine erscheinende Fluoreszenz
(FRET-Sonden) detektiert wird.
Der Trick der dPCR: das Ansatzvolumen der PCR wird in tausende (>20.000 je Target) separate
mikro-Volumina aufgeteilt. Dabei werden je Probe und je spezifischen
PCR-Target defacto abertausende PCRs parallel durchgeführt und durch
Detektion der Fluoreszenzen der Anteil der positiven PCRs bestimmt.
Da das Ergebnis je Mikro-PCR binär d. h. negativ oder positiv ist:
digitale PCR.